- 1. Cargar paquetes y base de datos
- 2. Algunas funciones para conocer/empezar a tratar los datos
- 3. Cambiar la naturaleza de las variables para empezar a trabajar con ellas
- 4. Renombrar variables
- 5. Tratamiento valores perdidos
- 6. Recodificar variables
- 7. Crear nuevas variables y fiabilidad de escalas
- 8. BUscar outliers y tratarlos
- 9. Aplicar filtros, criterios de exclusión…
Para cualquier error que detectéis o sugerencia, podéis escribir a jcolomer@ugr.es
1. Cargar paquetes y base de datos
library(pacman)
p_load(foreign, haven, dplyr, labelled, ggplot2, GGally,
ggridges, car, carData, viridis, viridisLite, hrbrthemes,
readr, tidyr, tibble, tidyverse, psych, sjmisc, install = TRUE)
data <- read_sav("C:/Users/User/Desktop/Tesis/Formación/Seminario R/Base de datos/CIS 2020 - Opinión pública y política fiscal/Database - CISPF.sav")2. Algunas funciones para conocer/empezar a tratar los datos
Con str(), estructure, podemos ver la estructura de nuestros datos
str(data) #Con str(), estructure, podemos ver la estructura de nuestros datos#str(data[1:3]) #vemos las 3 primeras variables (columnas)## tibble [2,926 x 3] (S3: tbl_df/tbl/data.frame)
## $ ESTU: dbl+lbl [1:2926] 3290, 3290, 3290, 3290, 3290, 3290, 3290, 3290, 3290,...
## ..@ label : chr "Nº de estudio"
## ..@ format.spss : chr "F4.0"
## ..@ display_width: int 4
## ..@ labels : Named num 3290
## .. ..- attr(*, "names")= chr "3290"
## $ CUES: dbl+lbl [1:2926] 2123, 680, 2522, 1090, 309, 1387, 487, 637, 937,...
## ..@ label : chr "Nº de cuestionario"
## ..@ format.spss : chr "F5.0"
## ..@ display_width: int 5
## ..@ labels : Named num [1:2926] 1 2 3 4 5 6 7 8 9 10 ...
## .. ..- attr(*, "names")= chr [1:2926] "1" "2" "3" "4" ...
## $ CCAA: dbl+lbl [1:2926] 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,...
## ..@ label : chr "Comunidad autónoma"
## ..@ format.spss : chr "F2.0"
## ..@ display_width: int 2
## ..@ labels : Named num [1:19] 1 2 3 4 5 6 7 8 9 10 ...
## .. ..- attr(*, "names")= chr [1:19] "Andalucía" "Aragón" "Asturias (Principado de)" "Balears (Illes)" ...
## - attr(*, "label")= chr "filelabel"names(data[5:10]) #Vemos los nombres de la variable 5 hasta la 10## [1] "MUN" "TAMUNI" "CAPITAL" "ZONA" "ENTREV" "TIPO_TEL"head(data[1:2]) #Vemos el nombre de la variable y las seis primeras puntuaciones (podemos pedir m?s en los argumentos de la funci?n)## # A tibble: 6 x 2
## ESTU CUES
## <dbl+lbl> <dbl+lbl>
## 1 3290 [3290] 2123 [2123]
## 2 3290 [3290] 680 [680]
## 3 3290 [3290] 2522 [2522]
## 4 3290 [3290] 1090 [1090]
## 5 3290 [3290] 309 [309]
## 6 3290 [3290] 1387 [1387]data$identificador <- 1:nrow(data)3. Cambiar la naturaleza de las variables para empezar a trabajar con ellas
class(data$P15) #Nos devuelve: [1] "haven_labelled" "vctrs_vctr" "double" ## [1] "haven_labelled" "vctrs_vctr" "double"table(data$P15) #Comprobamos como se distribuyen las respuestas###
## 1 2 8 9
## 453 2258 196 19Una forma rápida de pasar las variables de ‘haven labelled’ a numeric (más útil para la mayoría de nuestros análisis):
val_labels(data) <- NULLVolvemos a comprobar su naturaleza:
class(data$P15) #Nos devuelve: [1] "haven_labelled" "vctrs_vctr" "double" ## [1] "numeric"Y que no hayan cambiado los valores:
table(data$P15) #Comprobamos cómo se distribuyen las respuestas###
## 1 2 8 9
## 453 2258 196 19Una forma más tediosa pero en la vamos viendo qué hacemos paso a paso:
data$SEXO <- to_factor(data$SEXO)
data$EDAD <- as.numeric(data$EDAD)
data$ESCAFELI <- as.numeric(data$ESCAFELI)
data$ESCACONFIANZA <- as.numeric(data$ESCACONFIANZA)
data$P4_1 <- as.numeric(data$P4_1)
data$P4_2 <- as.numeric(data$P4_2)
data$P4_3 <- as.numeric(data$P4_3)
data$P4_4 <- as.numeric(data$P4_4)
data$P4_5 <- as.numeric(data$P4_5)
data$P4_6 <- as.numeric(data$P4_6)
data$P7 <- to_factor(data$P7)
data$ESCAIMPUESTOS <- as.numeric(data$ESCAIMPUESTOS)
data$INTERVENESTADO <- to_factor(data$INTERVENESTADO)
data$DESIGUALDAD <- as.numeric(data$DESIGUALDAD)
data$ESCAPOSICION <- as.numeric(data$ESCAPOSICION)
data$ESCIDEOL <- as.numeric(data$ESCIDEOL)
data$CLASESUB <- to_factor(data$CLASESUB)
data$INGREHOG <- as.numeric(data$INGREHOG)
data$NIVELESTENTREV <- as.numeric(data$NIVELESTENTREV)4. Renombrar variables
Con la función ‘rename’ del paquete ‘dplyr’, primero el nombre nuevo y después el nombre antiguo.
data <- data %>%
dplyr::rename(item1 = P4_1, item2 = P4_2, item3 = P4_3, item4 = P4_4,
item5 = P4_5, item6 = P4_6)5. Tratamiento valores perdidos
La forma más fácil y recomentable, utilizando el paquete ‘naniar’:
library(naniar)##
## Attaching package: 'naniar'## The following object is masked from 'package:sjmisc':
##
## all_nadata <- data %>%
replace_with_na(replace = list(ESCAFELI = c(98,99),
ESCACONFIANZA = c(98,99),
item1 = c(98,99),
item2 = c(98,99),
item3 = c(98,99),
item4 = c(98,99),
item5 = c(98,99),
item6 = c(98,99),
ESCAIMPUESTOS = c(98,99),
ESCAPOSICION = c(98,99),
CLASESUB = c(8, 9),
ESCIDEOL = c(97, 98, 99),
DESIGUALDAD = c(8, 9),
INTERVENESTADO = c(8, 9),
NIVELESTENTREV = c(16, 98, 99),
INGREHOG = c(98, 99)))Este mismo paquete nos permite visualizar la cantidad de valores perdidos por variable:
data %>%
dplyr::select(CLASESUB, ESCIDEOL, NIVELESTENTREV, INGREHOG) %>%
naniar::gg_miss_var()## Warning: It is deprecated to specify `guide = FALSE` to remove a guide. Please
## use `guide = "none"` instead.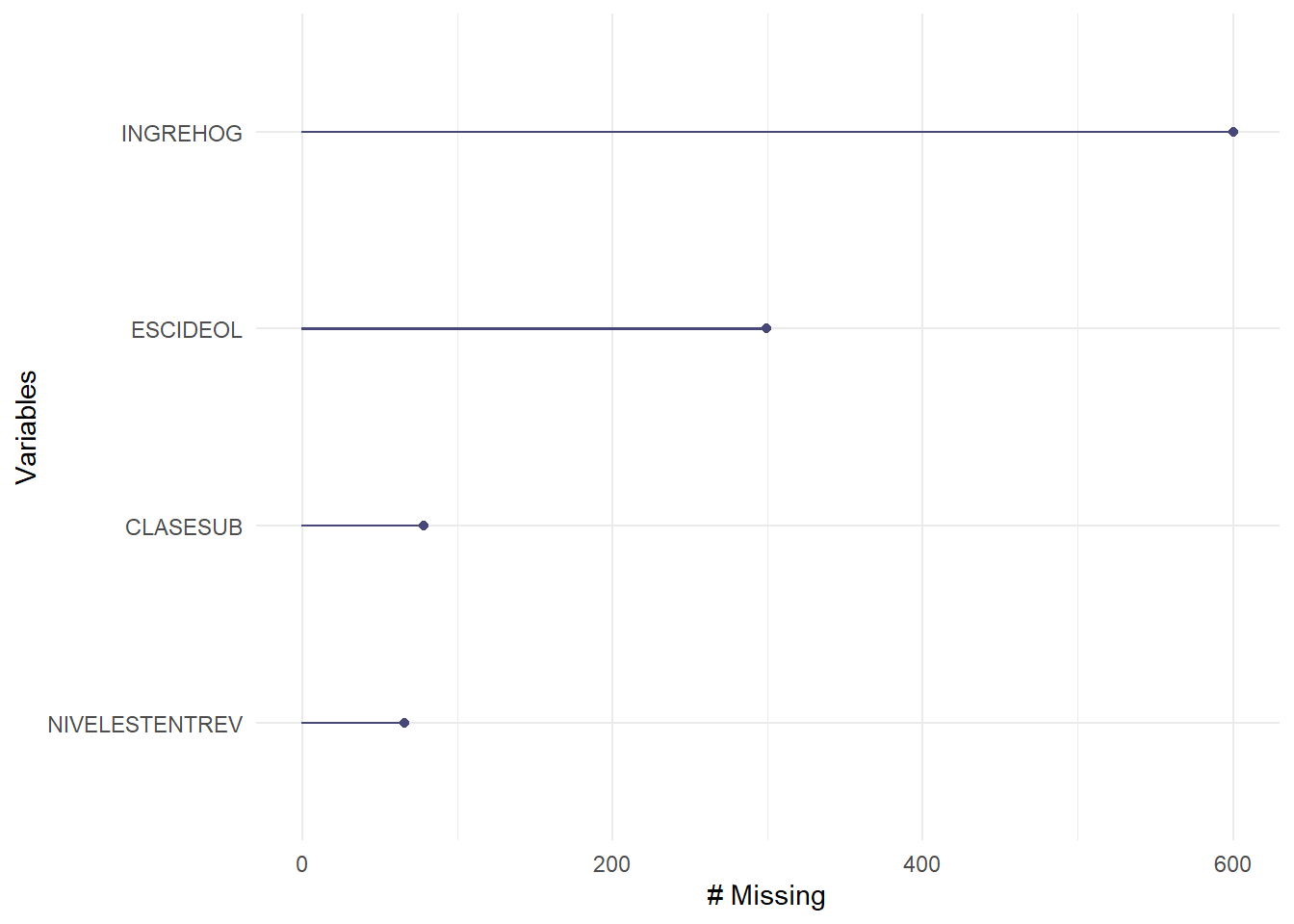
Una forma menos eficiente sería:
data$ESCAFELI <- na_if(data$ESCAFELI, '98')
data$ESCAFELI <- na_if(data$ESCAFELI, '99')
data$ESCACONFIANZA <- na_if(data$ESCACONFIANZA, '98')
data$ESCACONFIANZA <- na_if(data$ESCACONFIANZA, '99')6. Recodificar variables
data <- data %>%
mutate(item1rec = (10 - item1),
#Aqu? podr?amos a?adir todos los items de todas las escalas que quisi?semos
#o:
item2rec = dplyr::recode(item2, '0' = 10L, '1' = 9L, '2' = 8L,
'3' = 7L, '4' = 6L, '5' = 5L, '6' = 4L,
'7' = 3L, '8' = 2L, '9' = 1L,
'10' = 0L))7. Crear nuevas variables y fiabilidad de escalas
Podemos hacerlo así:
data$escala1a <- (data$item1 + data$item2 + data$item3 + data$item4 +
data$item5 + data$item6)/6Sin embargo, si hay algún valor perdido el resultado será Na. Así que una mejor forma de hacerlo, que lleva a cabo el cálculo sin tener en cuenta los valores perdidos, sería la siguiente:
data$escala1b <- rowMeans(data[,c("item1", "item2", "item3",
"item4", "item5", "item6")], na.rm=TRUE)También podemos calcular la fiabilidad de la escala que acabamos de crear:
data %>%
dplyr::select(item1, item2, item3, item4, item5, item6) %>%
alpha(check.keys = TRUE)## Number of categories should be increased in order to count frequencies.##
## Reliability analysis
## Call: alpha(x = ., check.keys = TRUE)
##
## raw_alpha std.alpha G6(smc) average_r S/N ase mean sd median_r
## 0.61 0.67 0.64 0.25 2 0.011 8.9 1 0.23
##
## lower alpha upper 95% confidence boundaries
## 0.59 0.61 0.63
##
## Reliability if an item is dropped:
## raw_alpha std.alpha G6(smc) average_r S/N alpha se var.r med.r
## item1 0.60 0.66 0.63 0.28 1.9 0.011 0.0088 0.30
## item2 0.62 0.65 0.61 0.27 1.8 0.011 0.0078 0.23
## item3 0.52 0.61 0.58 0.24 1.6 0.013 0.0105 0.22
## item4 0.53 0.60 0.56 0.23 1.5 0.013 0.0081 0.21
## item5 0.57 0.61 0.57 0.24 1.6 0.012 0.0067 0.23
## item6 0.57 0.61 0.57 0.23 1.5 0.012 0.0078 0.21
##
## Item statistics
## n raw.r std.r r.cor r.drop mean sd
## item1 2905 0.50 0.53 0.35 0.26 8.6 1.56
## item2 2891 0.72 0.56 0.41 0.35 7.8 2.78
## item3 2869 0.67 0.63 0.52 0.45 9.0 1.82
## item4 2907 0.63 0.67 0.57 0.44 9.0 1.51
## item5 2908 0.54 0.63 0.53 0.36 9.2 1.27
## item6 2918 0.52 0.65 0.55 0.40 9.5 0.888. BUscar outliers y tratarlos
Para hacerlo visualmente, aunque puede ser poco informativo en algunos casos:
boxplot(data$EDAD)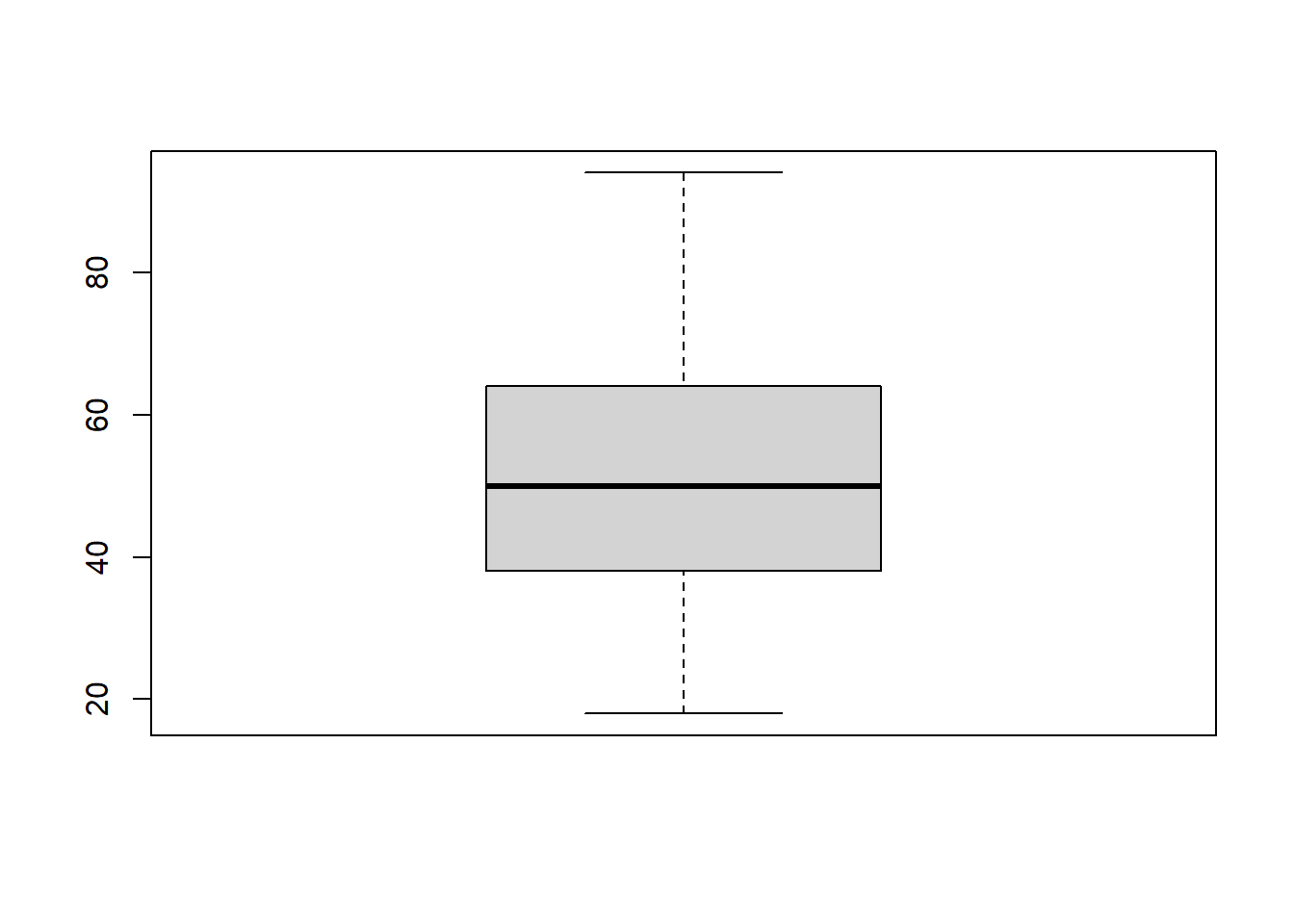
Otra forma sería la siguiente, con el paquete ‘performance’:
performance::check_outliers(data$EDAD)## Warning: 21 outliers detected (cases 438, 563, 566, 710, 1119, 1136, 1317, 1337, 1412, 1492, 1705, 2263, 2265, 2321, 2418, 2482, 2541, 2551, 2552, 2673, 2709).Si elegimos esta forma hay que tener en cuenta el método (podemos elegirlo) y ver bien las especificaciones del paquete.
Para ver los valores concretos de los outliers, utilizar este código:
data[438, "EDAD"] ## # A tibble: 1 x 1
## EDAD
## <dbl>
## 1 90data[563, "EDAD"]## # A tibble: 1 x 1
## EDAD
## <dbl>
## 1 88data[566, "EDAD"]## # A tibble: 1 x 1
## EDAD
## <dbl>
## 1 90Una forma más eficiente para, por ejemplo, ver la edad y el nº identificador de los casos que tienen más de 87 años:
data %>%
filter(EDAD > 87) %>%
dplyr::select(EDAD, identificador)## # A tibble: 21 x 2
## EDAD identificador
## <dbl> <int>
## 1 90 438
## 2 88 563
## 3 90 566
## 4 88 710
## 5 89 1119
## 6 90 1136
## 7 88 1317
## 8 94 1337
## 9 89 1412
## 10 90 1492
## # ... with 11 more rows9. Aplicar filtros, criterios de exclusión…
Podemos aplicar filtros con la función filter(), como acabamos de hacer:
data %>%
filter(EDAD < 87) %>%
dplyr::select(ESCAIMPUESTOS, ESCAPOSICION, EDAD, DESIGUALDAD, ESCIDEOL) %>%
sjPlot::tab_corr()## Registered S3 methods overwritten by 'parameters':
## method from
## as.double.parameters_kurtosis datawizard
## as.double.parameters_skewness datawizard
## as.double.parameters_smoothness datawizard
## as.numeric.parameters_kurtosis datawizard
## as.numeric.parameters_skewness datawizard
## as.numeric.parameters_smoothness datawizard
## print.parameters_distribution datawizard
## print.parameters_kurtosis datawizard
## print.parameters_skewness datawizard
## summary.parameters_kurtosis datawizard
## summary.parameters_skewness datawizard| ESCAIMPUESTOS | ESCAPOSICION | EDAD | DESIGUALDAD | ESCIDEOL | |
|---|---|---|---|---|---|
| ESCAIMPUESTOS | -0.034 | -0.028 | -0.121*** | 0.333*** | |
| ESCAPOSICION | -0.034 | -0.139*** | 0.072*** | -0.128*** | |
| EDAD | -0.028 | -0.139*** | 0.095*** | 0.040* | |
| DESIGUALDAD | -0.121*** | 0.072*** | 0.095*** | -0.171*** | |
| ESCIDEOL | 0.333*** | -0.128*** | 0.040* | -0.171*** | |
| Computed correlation used pearson-method with listwise-deletion. | |||||
O directamente crear una nueva base de datos aplicando nuestros criterios de exclusión:
data2 <- subset(data[!(data$SEXO=="2" | is.na(data$ESCAIMPUESTOS)
| data$EDAD>= 87),])En este caso habríamos eliminado a las mujeres (SEXO = 2), las personas con valores perdidos en la variable ESCAIMPUESTOS y las personas que tengan 87 años y más.